ВВЕДЕНИЕ
Заболевание COVID-19 связано со специфичной клинической картиной вследствие неэффективного ответа иммунной системы и высокого уровня провоспалительных цитокинов (включая интерлейкины 1, 6, 4, 10 и интерферон-гамма), известного как «цитокиновый шторм» [1]. Такой гипервоспалительный иммунологический ответ, вызванный инфекцией SARS-CoV-2, является результатом начального включения врожденного иммунного ответа, который формирует первую линию защиты от патогенов и, в свою очередь, стимулирует активацию адаптивного иммунитета [2]. По этой причине для достижения эффективного контроля над инфекцией крайне важно, чтобы иммунологический ответ был сбалансирован; это позволяет избежать как чрезмерного воспаления, оказывающего повреждающее действие (что наблюдается в легких пациентов с COVID-19), так и низкой активации иммунной системы, способствующей распространению вируса [3, 4].
Врожденный иммунитет идентифицирует компоненты вирусов («патоген-ассоциированные молекулярные паттерны», PAMP) с помощью паттерн-распознающих рецепторов (PRR) [5]. Взаимодействие PAMPs-PRR приводит к внутриклеточному сигнальному каскаду, необходимому как для реализации противовирусной активности за счет продукции интерферонов, так и для активации иммунной системы за счет секреции цитокинов [5]. Семейство PRR включает различные компоненты [6], участвующие в распознавании вирусных РНК-инфекций, такие как RIG-I-подобные рецепторы (RLR), например, RIG-I и MDA5, и Toll-подобные рецепторы (TLRs) [7]. Само по себе семейство Toll-подобных рецепторов (TLR) представляет собой мембраносвязанные PRR, которые обнаруживают молекулярные паттерны, связанные с вирусами, бактериями и грибами, на плазматической мембране и внутри эндосом [8, 9]. Миелоидная дифференцировка первичного ответа 88 (MyD88) и TIR-домен, который содержит адаптор, индуцирующий интерферон-бета (TRIF, также известный также как TICAM1), служат двумя основными путями передачи сигналов TLRs [10]. Белки TRAF и IRAK в сигнальных путях вызывают активацию ядерного фактора-kB (NF-kB) и регуляторного фактора интерферона (IRF), что, в свою очередь, приводит к продукции интерферонов (ИФН) 1-го типа и провоспалительных цитокинов, таких как интерлейкин 1 (ИЛ-1), ИЛ-6, фактор некроза опухоли-α (ФНО-α) и ИЛ-12 [11]. Например, TLR3 участвует в обнаружении многих РНК-вирусов и в изменении патогенеза заболеваний дыхательных путей, возникающих в результате респираторных вирусных инфекций, таких как IAV, респираторно-синцитиальный вирус (RSV) и риновирусные инфекции [12, 13]. Базальные уровни экспрессии TLR3 обнаруживаются в тканях легких (альвеолярных клетках человека и бронхиальных эпителиальных клетках), а также в различных других популяциях иммунных клеток [14]. В клетках TLR3 прикрепляется к мембране эндосом, где распознает мотивы двухцепочечной РНК (дцРНК) от патогенов [15]. После связывания мотива дцРНК рецептор TLR3 димеризуется и рекрутирует адапторный белок TRIF [16]. Рекрутирование TRIF в эндосомы влечет за собой передачу сигналов для активации факторов транскрипции, включая IRF3 и NF-kB [17].
TLR7/8 представляют собой тандемно дуплицированные гены на Х-хромосоме, которые расположены в мембране эндосомы и распознают одноцепочечную РНК, и синтетические олигорибонуклеотиды, такие как имидазохинолин, имихимод и R-848 [18, 19]. Следовательно, они могли участвовать в распознавании генома SARS-CoV-2 [20]. Связывание поверхностного гликопротеина S на оболочке вируса можно обнаружить с помощью TLR7. TLR7 экспрессируется на моноцитах-макрофагах и дендритных клетках, и его активация вызывает продукцию ИЛ-1, ИЛ-6, моноцитарного хемоаттрактантного белка-1, MIP-1A, ФНО-α и ИФН 1-го типа [21]. Плазматические дедритные клетки обнаруживают вирусную оцРНК через эндосомальный рецептор TLR [22], активируемый геномными фрагментами, богатыми гуанином и урацилом (богатые ГУ), полученными в результате эндосомального процессинга вируса независимо от инфекции [23]. Кроме того, у вирусов одноцепочечной РНК, таких как SARS-CoV-2, можно наблюдать зависящий от пола ответ, поскольку ген TLR7/8 находится на Х-хромосоме [4]. Гиперэкспрессия TLR7 может привести к более легкому протеканию заболевания, вызванного вирусом с одноцепочечной РНК, вследствие более высокого иммунного ответа. Полногеномное секвенирование SARS-CoV, MERS-CoV и SARS-CoV-2 показало, что TLR7 может быть в большей степени вовлечен в патогенез инфекции, вызванной SARS-CoV-2, по сравнению с SARS-CoV и MERS-CoV, поскольку возбудитель новой коронавирусной инфекции содержит больше одноцепочечных РНК-мотивов, способных связываться с TLR7 [13]. Можно говорить о значительной роли TLR7 в противовирусном врожденном ответе на SARS-CoV- 2 [13]. Помимо этого, TLRs косвенно участвует в адаптивной иммунной системе, контролируя экспрессию ко-стимулирующих молекул [3].
Как только вирусные РНК-сенсоры активированы, нижестоящая сигнализация задействуется, чтобы индуцировать факторы транскрипции в ядре, которые, в свою очередь, способствуют экспрессии генов-мишеней, включая ИФН 1-го и 3-го типов, а также ряд других важных провоспалительных цитокинов [24]. Среди задействованных транскрипционных факторов ключевую роль играют IRF3 и NF-kB [22], при этом белок IRF3 участвует в продукции ИФН [25], тогда как NF-κB в основном используется для индукции провоспалительного ответа [23]. При этом, даже если и IRF3, и NF-κB имеют решающее значение в передаче сигналов, воспринимающих вирусную РНК, они по-разному индуцируются эндосомальными TLR-3 и -7 [26, 27]. Фактически, в то время как активация TLR7 приводит в основном к рекрутированию NF-kB, TLR3 обычно активирует как NF-kB, так и сигнал IRF3 [17]. Эта дифференциальная передача сигналов возможна, потому что и TLR3, и TLR7 включают киназу TBK1, которая отвечает за фосфорилирование IRF3 и NF-κB. За этим первым сигналом следует второй, адресованный всем окружающим клеткам, которые вынуждены экспрессировать большое количество ИФН-стимулируемых генов, что в итоге приводит к элиминации вируса из организма [16].
Целью настоящей работы стало исследование профиля экспрессии и молекулярных механизмов распознающих молекул врожденного иммунитета в ответ на вирус SARS CoV-2, что в дальнейшем может быть использовано как один из способов контроля иммунной системы в ответ на действие данного возбудителя.
МАТЕРИАЛ И МЕТОДЫ
В исследование были включены пациенты (n=26), переболевшие тяжелой формой COVID- 19. Контрольную группу (n=10) составили условно здоровые лица, контактировавшие с инфекцией SARS CoV-2, но без подтвержденных результатов теста на SARS CoV-2 и без клинической симптоматики.
Критерии исключения из исследовательского протокола были следующими:
- сопутствующие и хронические заболевания (легочные – муковисцидоз, абсцесс легких, эмпиема плевры, активный туберкулез; внелегочные – застойная сердечная недостаточность, острая/хроническая печеночная недостаточность, острая почечная недостаточность/хроническая болезнь почек, злокачественные образования, иммунодефициты различной этиологии);
- наличие в анамнезе положительной реакции на антигены ВИЧ-инфекции (Retroviridae, Orthoretrovirinae, Lentivirus, Human immunodeficiency virus), гепатитов В (Hepadnaviridae, Orthohepadnavirus, Hepatitis B virus) и С (Flaviviridae, Hepacivirus, Hepatitis С virus);
- наличие иных (лабораторно подтвержденных) острых инфекционных и/или неинфекционных заболеваний на момент включения в протокол;
- применение (в течение свыше 14 сут) иммунодепрессантов или других иммуномодулирующих препаратов на протяжении 6 мес, предшествовавших исследованию;
- протекающая беременность или лактация.
Всем пациентам с новой коронавирусной инфекцией проводилось комплексное клиническое обследование, включавшее компьютерную томографию (КТ) органов грудной клетки, пульсоксиметрию и лабораторные тесты на наличие РНК (антигена) SARS-CoV-2. В работе использовался биоматериал в виде соскобов слизистых оболочек верхних дыхательных путей (ротоглотки, носоглотки), а также ротовой полости. Забор материала выполнялся цитощеточкой типа D (производитель – «Юнона», Россия) и транспортировался в лабораторию в пробирке на 1,5 мл в физиологическом растворе («Панеко», Россия) в закрытом пакете.
Исследование проводилось при информированном согласии пациентов.
Из полученного биоматериала выделяли РНК методом сорбции на силикагеле с использованием коммерческого набора «РИБО-сорб» (Amplisense, Россия) в соответствии с протоколом проведения для этого набора. Проверка качества выделенной РНК для исследования экспрессии генов TLR3, TLR7 осуществлялась с помощью спектрофотометра Nanodrop 2000® (Thermo Scientific, США). Реакцию обратной транскрипции и последующую полимеразную цепную реакцию в реальном времени (ПЦР-РВ) проводили с помощью коммерческих наборов «Набор для обратной траскрипции ОТ-1», «Набор реагентов для проведения ПЦР-РВ в присутствии красителя SYBR Green I» («Синтол», Россия). Последовательности праймеров для генов TLR3 и TLR7 подбирали с применением программы Vector NTI, анализируя последовательность интересующих генов, полученную из GenBank. Стандартизация результатов ПЦР-РВ выполнялась по уровню экспрессии гена β-актина. Обхват экспрессии гена был посчитан методом относительного анализа экспрессии гена с использованием метода ∆∆Ct.
Статистическая обработка результатов осуществлялась с использованием непараметрических критериев Манна–Уитни при помощи программы GraphPad v.8.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Поскольку вирус SARS-CoV-2 преимущественно поражает слизистую верхних дыхательных путей, именно на уровне слизистых оболочек происходит распознавание вирусных частиц рецепторами врожденного иммунитета. Нам было интересно посмотреть, как на уровне слизистой ротоглотки, носоглотки и ротовой полости изменяется экспрессия распознающих рецепторов в ответ на вирусную инвазию.
Результаты изменения экспрессии генов TLR3, TLR7 в слизистых оболочках верхних дыхательных путей, полученные в нашей работе, отражены на рисунках 1–3. На уровне слизистых оболочек носоглотки в период протекания COVID-19 было установлено снижение экспрессионного профиля врожденных молекул распознавания TLR3 и TLR7. У пациентов с тяжелым течением новой коронавирусной инфекции на 1-е, 15-е и 30-е сутки заболевания уровень экспрессии TLR7 был достоверно ниже в 42, 128 и 26 раз соответственно относительно группы условно здоровых лиц. Помимо этого, у пациентов с тяжелым COVID-19 по сравнению с контрольной группой на 15-е и 30-е сутки наблюдалось достоверное снижения уровня экспрессии TLR3 в 151 и 38 раз соответственно (см. рис. 1).

На уровне слизистых оболочек ротоглотке нами также было обнаружено снижение уровня экспрессии TLR3 и TLR7 на протяжении заболевания COVID-19. Так, уровень экспрессии TLR7 у исследованных пациентов в дебюте (1-е сутки) заболевания оказался в 32 раза ниже, чем в группе контроля. Что касается экспрессии гена TLR3, то у пациентов с тяжелой формой новой коронавирусной инфекции на 1-е, 15-е и 30-е сутки заболевания она была достоверно ниже в 1,3×103, 4,13×105 и 1,88×105 раз соответственно относительно условно здоровых лиц (рис. 2).
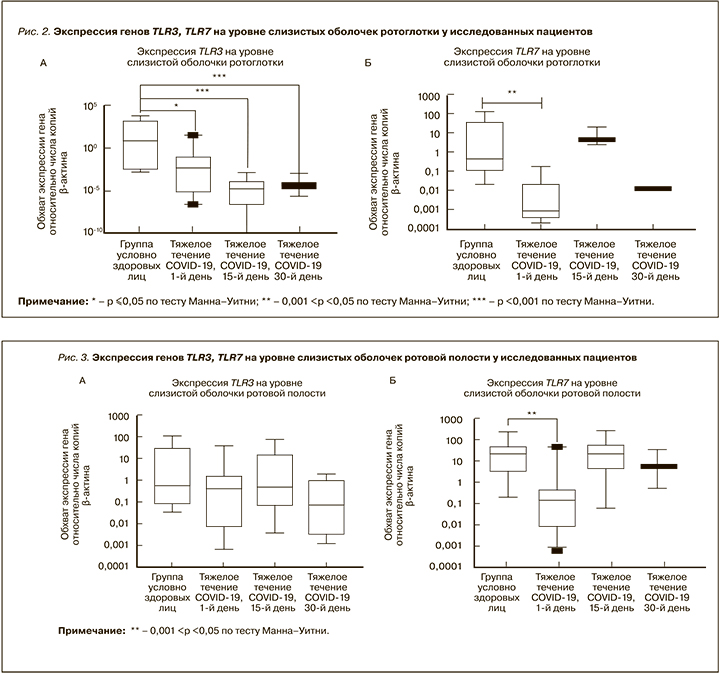
На уровне эпителия слизистой оболочки ротовой полости у лиц с тяжелым течением COVID-19 было установлено достоверное снижение уровня экспрессии гена TLR7 в 1,9×102 раз в дебюте заболевания (1-й день) в сравнении с группой условно здоровых лиц. Что касается экспрессии TLR3, то в основной группе пациентов была выявлена тенденция к снижению этого показателя в 1-е сутки, однако на 15-е сутки от начала заболевания у больных с тяжелым течением COVID-19 уровень экспрессии этого гена был сравнимым с таковым в контроле, а на 30-е сутки – незначительно сниженным (рис. 3).
Исходя из литературных данных, сниженный экспрессионный профиль распознающих молекул врожденного иммунитета, таких как TLR3, TLR7, на уровне эпителиальных клеток верхних дыхательных путей может быть связан с действием гена ORF9b вируса SARS-CoV-2. Han L. et al. показали, что этот ген ингибирует на молекулярном уровне передачу сигнала по пути TLR3-TRIF и тем самым снижает противовирусный иммунологический ответ, способствуя вирусной репликации [28]. Поскольку TLR7 распознает одноцепочечную РНК (оцРНК), увеличение его экспрессии в клетках слизистой оболочки верхних дыхательный путей в дебюте заболевания у лиц с тяжелым течением COVID-19 может указывать на инфицирование другими штаммами вируса, содержащими оцРНК [21]. В соответствии с иммуноинформационным подходом геном SARS-CoV-2 содержит больше фрагментов оцРНК, которые распознаются TLR7/8, что свидетельствует о гиперактивации врожденного иммунитета на SARS-CoV-2 с индукцией эффективного провоспалительного ответа.
ЗАКЛЮЧЕНИЕ
В нашем исследовании наблюдалось снижение экспрессионного профиля врожденных молекул распознавания на уровне слизистых оболочек верхних дыхательных путей в ответ на инвазию вируса SARS CoV-2. Такие изменения в уровне экспрессии генов могут говорить об изменении вирусными частицами сигнальных, молекулярных и биохимических путей передачи сигнала от рецептора к продуктам реакции; следовательно, можно предположить, что у лиц с такой гипоэкспрессией развиваются как тяжелое течение заболевания COVID-19, так и «постковидный» синдром после выздоровления. Кроме того, подобные изменения уровня экспрессии гена распознающих молекул врожденного иммунитета могут быть следствием генетических вариантов полиморфизма гена, что наводит на мысль о необходимости генетического скрининга в целях предотвращения тяжелого течения COVID-19.



